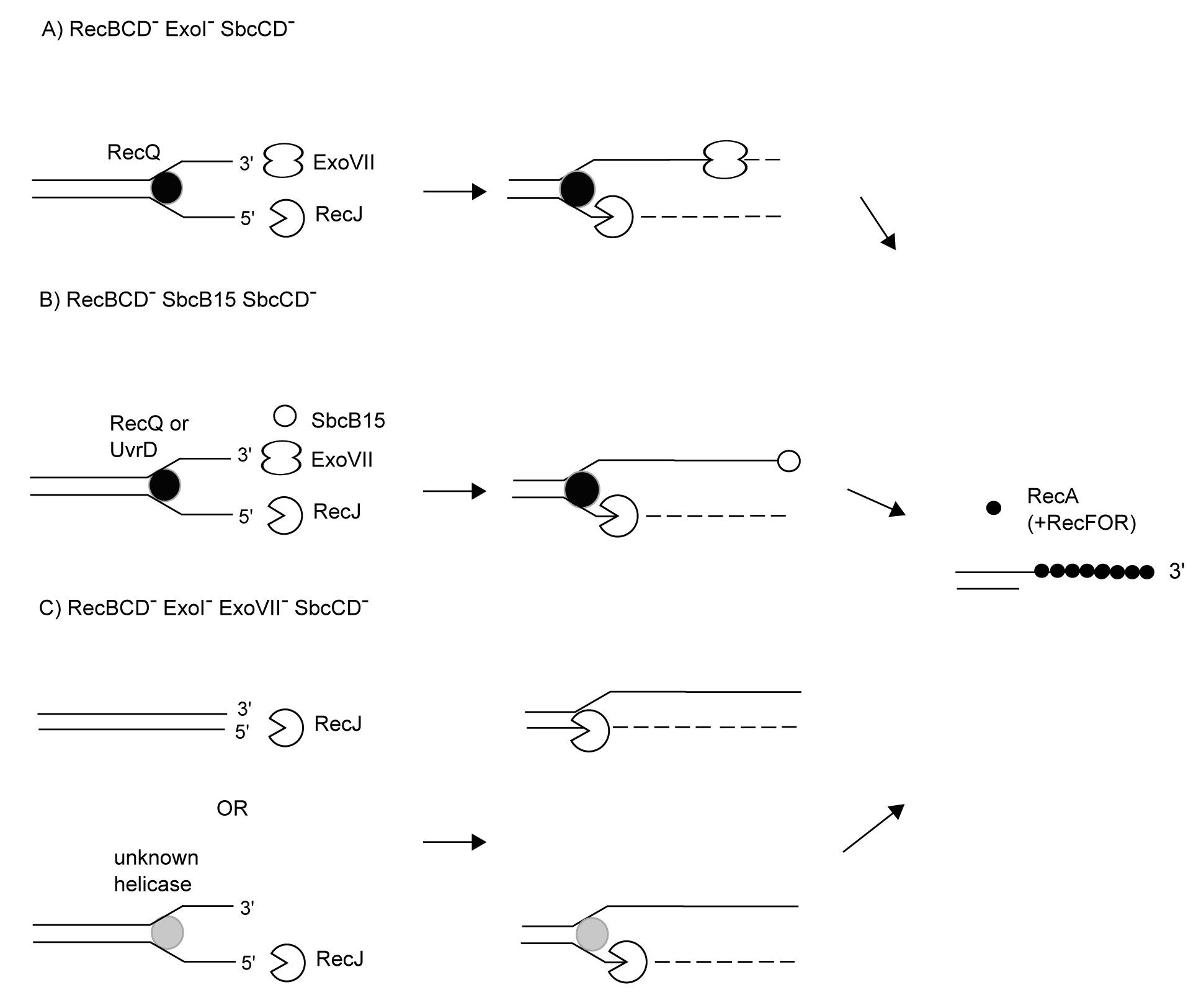
Homologna rekombinacija je esencijalan biološki proces koji sudjeluje u popravku DNA i u očuvanju integriteta genoma. Ključnu ulogu u rekombinaciji kod bakterija ima protein RecA, a njegovi homolozi prisutni su i u viših organizama, uključujući čovjeka. Protein RecA djeluje u obliku nukleoproteinskog filamenta koji se formira na jednolančanoj DNA te katalizira sparivanje i izmjenu lanaca između dva homologna dupleksa. U bakteriji Escherichia coli, u sklapanju RecA filamenta sudjeluju proteinski kompleksi RecBCD i RecFOR. Nedavno smo otkrili da neki mutanti bakterije E. coli mogu relativno dobro rekombinirati i u odsutnosti proteinskih kompleksa RecBCD i RecFOR. Ta rekombinacija ovisi o proteinu RecA što ukazuje na postojanje alternativnog mehanizma stvaranja RecA filamenta. Cilj ovog projekta je genetički karakterizirati ovaj RecBCD- RecFOR-neovisan put rekombinacije te rasvjetliti novi mehanizam sklapanja RecA filamenta u bakteriji E. coli.
Iako je protein RecA ključan za rekombinaciju u bakterija, recA mutanti radiorezistentne bakterije Deinococcus radiodurans sposobni su u određenoj mjeri za rekombinacijski popravak dvolančanih lomova DNA. Naši nedavni rezultati pokazali su da je taj RecA-neovisan popravak neprecizan i da dovodi do velikih kromosomskih rearanžmana. U sklopu projekta nastojat ćemo otkriti ključne gene/proteine uključene u RecA-neovisni popravak DNA te karakterizirati, na razini sekvence, velike genomske rearanžmane u bakteriji D. radiodurans.
S obzirom da je rekombinacija fundamentalan proces sačuvan tijekom evolucije, naša istraživanja na bakterijama mogla bi pomoći u rasvjetljavanju rekombinacijskih mehanizama u višim, eukariotskim organizmima. Rezultati ovog projekta mogli bi stoga pomoći i u razumijevanju mehanizama nastanka raka i nasljednih bolesti kod čovjeka koje su povezane s poremećajima u rekombinacijskim procesima.
Publikacije
Zahradka K, Repar J, Đermić D, Zahradka D. Chromosome Segregation and Cell Division Defects in Escherichia coli Recombination Mutants Exposed to Different DNA-Damaging Treatments // Microorganisms, 11 (2023), 3; 11030701, 30. doi: 10.3390/microorganisms11030701
Zahradka K, Repar J, Đermić D, Zahradka D. Genetic analysis of transductional recombination in Escherichia coli reveals differences in the postsynaptic stages of RecBCD and RecFOR pathways // Periodicum Biologorum, 124 (2022), 3-4; 97-106. doi: 10.18054/pb.v124i3-4.23604
Repar J, Zahradka D, Sović I, Zahradka K. Characterization of gross genome rearrangements in Deinococcus radiodurans recA mutants // Scientific Reports, 11 (2021), 1; 10939, 11. doi: 10.1038/s41598-021-89173-9
Buljubašić M, Hlevnjak A, Repar J, Đermić D, Filić V, Weber I, Zahradka K, Zahradka D. RecBCD- RecFOR-independent pathway of homologous recombination in Escherichia coli // DNA Repair, 83 (2019), 102670, 16. doi: 10.1016/j.dnarep.2019.102670
Feliciello I, Zahradka D, Zahradka K, Ivanković S, Puc N, Đermić D. RecF, UvrD, RecX and RecN proteins suppress DNA degradation at DNA double-strand breaks in Escherichia coli // Biochimie, 148 (2018), 116-126. doi: 10.1016/j.biochi.2018.03.005
Đermić E, Zahradka D, Vujaklija D, Ivanković S, Đermić D. 3'-terminated Overhangs Regulate DNA Double-Strand Break Processing in Escherichia coli // G3-Genes Genomes Genetics 7 (2017), 9; 3091-3102. doi: 10.1534/g3.117.043521
Ivanković S, Vujaklija D, Đermić D. Nucleolytic degradation of 3′-ending overhangs is essential for DNA-end resection in RecA-loading deficient recB mutants of Escherichia coli // DNA Repair, 57 (2017), 56-65. doi: 10.1016/j.dnarep.2017.06.024
Repar J, Supek F, Klanjšček T, Warnecke T, Zahradka K, Zahradka D. Elevated rate of genome rearrangements in radiation-resistant bacteria // Genetics, 205 (2017), 4; 1677-1689. doi: 10.1534/genetics.116.196154
Đermić D. Double-strand break repair mechanisms in Escherichia coli: Recent insights // Advances in Genomics and Genetics, 5 (2015), 35-42. doi: 10.2147/AGG.S51699
Diplomski rad
Hleb A. Genomski rearanžmani u bakteriji Deinococcus radiodurans: uloga proteina RecG / Hulak, Nataša ; Zahradka, Ksenija (mentor); Zagreb, Agronomski fakultet, 2018.
Sudjelovanja na konferencijama/ljetnim školama
Repar J. Popravak dvolančanih lomova DNA i stabilnost genoma u bakteriji Deinococcus radiodurans // DNA dvostruka uzvojnica – sedamdeset godina kasnije, CePOZiR & HBD stručni skup Zagreb, Hrvatska, 28.11.2023. (pozvano predavanje)
Zahradka K, Repar J, Đermić D, Zahradka D. Chromosome segregation and cell division defects in Escherichia coli after induction of double- strand DNA breaks // Power of Microbes in Industry and Environment 2023 : Book of Abstracts / Teparić R, Leboš Pavunc A, Kifer D (ur.). Zagreb: Hrvatsko mikrobiološko društvo, 2023. str. 73-73 (poster)
Zahradka D. RecBCD- RecFOR-independent pathway of homologous recombination in Escherichia coli // FEMS Summer School for Postdocs. Split, Hrvatska 28.08.2019-07.09.2019 (pozvano predavanje i mentorstvo radne grupe)
Buljubašić M, Zahradka K, Hlevnjak A, Repar J, Đermić D, Zahradka D. RecBCD- RecFOR-independent pathway of homologous recombination in Escherichia coli // 4th Congress of Croatian Geneticists with International Participation - Book of Abstracts / Šarčević H, Ugarković Đ, Vujaklija D et al. (ur.). Zagreb: Hrvatsko genetičko društvo, 2018. str. 25-25 (predavanje)
Đermić E, Zahradka D, Vujaklija D, Đermić D. A new role of RecA protein in Escherichia coli? // 4th Congress of Croatian Geneticists with International Participation : Book of Abstracts / Šarčević H, Ugarković Đ, Vujaklija D et al. (ur.). Zagreb: Hrvatsko genetičko društvo, 2018. str. 44-44 (poster)
Repar J, Supek F, Klanjscek T, Warnecke T, Zahradka K, Zahradka D. Genome rearrangements in radiation-resistant bacteria // 4th Congress of Croatian Geneticists with International Participation : Book of Abstracts / Šarčević H, Ugarković Đ, Vujaklija D et al. (ur.). Zagreb: Hrvatsko genetičko društvo, 2018. str. 24-24 (predavanje)
Đermić E, Zahradka D, Vujaklija D, Ivanković S, Đermić D. 3'-terminal Overhangs Regulate DNA Double-Strand Break Processing in Escherichia coli // EMBO Conference on Meiosis 2017 : Book of Abstracts / Tolić I (ur.). 2017. str. 93-93 (poster)
Hlevnjak A, Paradžik T, Kazazić S, Filić Ž, Zahradka D, Vujaklija D. Phosphorylation of the SSB protein from E. coli // John Innes/Rudjer Bošković Summer School in Applied Molecular Microbiology: Microbial Diversity and Specialised Metabolites. Dubrovnik, Hrvatska, 10.09.2016-18.09.2016 (poster)
Repar J. Elevated rate of genome rearrangements in radiation resistant bacteria // CEGSDM 2016 - Central European Genome Stability and Dynamics Meeting. Zagreb, Hrvatska, 15.10.2016-16.10.2016 (pozvano predavanje)
Zahradka K, Hlevnjak A, Repar J, Buljubašić M, Zahradka D. Genetic differences in postsynaptic stages of two recombinational repair pathways in Escherichia coli // EEMGS 2016 Annual Meeting - Programme & Abstract book / EEMGS (ur.). Kopenhagen, 2016. str. 146-146 (poster)